¡Tu carrito está actualmente vacío!
El Código Secreto del Crecimiento Bacteriano: Descubren un “Plan Maestro” Universal en Cómo las Bacterias Usan sus Proteínas
El Dilema de Crecer Rápido o Ser Eficiente
Las bacterias, a pesar de su aparente simplicidad, son maestras de la adaptación y la eficiencia. Para sobrevivir y prosperar en entornos cambiantes, deben tomar decisiones cruciales sobre cómo utilizar sus recursos limitados. Una de las decisiones más importantes es cómo “asignar” su proteoma, es decir, qué proteínas producir y en qué cantidad. Esto es especialmente crítico para el crecimiento: ¿deben invertir masivamente en la maquinaria para fabricar más proteínas (ribosomas) para crecer rápido, o ser más conservadoras y versátiles? Durante mucho tiempo, nuestro entendimiento de estas estrategias se basó en estudios extensivos del organismo modelo Escherichia coli (E. coli). Sin embargo, ¿son estas reglas aplicables a otras bacterias, especialmente a aquellas que crecen a velocidades vertiginosas? Un nuevo e intrigante estudio publicado en PNAS por Manlu Zhu, Matteo Mori, Terence Hwa y Xiongfeng Dai, desafía algunas nociones comunes y revela la existencia de un “plan maestro” o “anteproyecto” sorprendentemente conservado en la asignación del proteoma que trasciende grandes distancias filogenéticas.
Más Allá de E. coli: Estudiando a los “Fórmula 1” del Mundo Microbiano
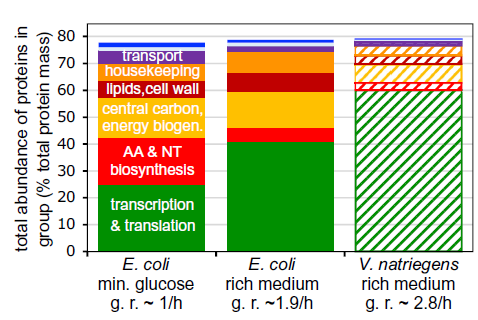
El equipo de investigación se centró en comparar a E. coli y Bacillus subtilis (otro modelo bien estudiado, pero filogenéticamente distante) con Vibrio natriegens, una bacteria marina conocida por ser una de las de más rápido crecimiento en el planeta, capaz de duplicarse en menos de 10 minutos en condiciones óptimas. La pregunta era: ¿cómo logra V. natriegens esta hazaña? ¿Simplemente produce muchos más ribosomas que E. coli?
Mediante experimentos cuantitativos exhaustivos, midiendo la composición completa del proteoma (el conjunto de todas las proteínas) y la cinética de los ribosomas (la velocidad a la que fabrican nuevas proteínas) en estas tres especies bajo diversas condiciones nutricionales y temperaturas, los investigadores descubrieron patrones sorprendentes.
La Sorpresa: La Rigidez del Plan y la Flexibilidad de las Enzimas
Contrario a la intuición, V. natriegens no simplemente dedica una porción mucho mayor de su proteoma a los ribosomas en comparación con E. coli para crecer más rápido. Los hallazgos clave fueron:
- Un Programa de Asignación Invariante: A pesar de las enormes diferencias en las tasas de crecimiento y la lejanía evolutiva, las tres especies bacterianas mostraron un programa de asignación del proteoma notablemente rígido y conservado cuando se las “normalizaba” adecuadamente. No es la tasa de crecimiento absoluta la que dicta directamente cómo se distribuyen las proteínas.

- La Métrica Universal: Saturación del Ribosoma: Los investigadores descubrieron que la clave para entender esta asignación conservada es una métrica interna: el grado de “saturación” del ribosoma. Esto se refiere a cuán ocupados están los ribosomas fabricando proteínas, lo cual está directamente relacionado con la velocidad de elongación (la rapidez con la que los ribosomas añaden aminoácidos a una cadena proteica). Cuando se escalan los datos de asignación del proteoma (por ejemplo, la fracción de proteínas dedicada a la biosíntesis de aminoácidos o al metabolismo central) en función de esta métrica de saturación ribosomal (o su equivalente, la velocidad de elongación normalizada por la máxima posible a esa temperatura), ¡los datos de las tres especies colapsan en curvas universales!

- La Flexibilidad Reside en la Cinética Enzimática: Si el “plan” de asignación de proteínas es tan rígido, ¿cómo crece V. natriegens mucho más rápido? La respuesta está en la cinética de sus enzimas. Los ribosomas de V. natriegens (y presumiblemente muchas otras de sus enzimas) son intrínsecamente más rápidos. Es como si V. natriegens estuviera operando a una “temperatura efectiva” más alta que E. coli o B. subtilis. De hecho, V. natriegens a 30°C se comporta de manera muy similar a E. coli a 37°C en términos de velocidad de elongación ribosomal y asignación del proteoma.

Desafiando Nociones Evolutivas Comunes
Estos hallazgos son significativos porque desafían la idea común de que la expresión génica (y por ende, la composición del proteoma) evoluciona más rápidamente que la función intrínseca de las proteínas (su cinética). Aquí se observa lo contrario: el “plan” de cómo se distribuyen las proteínas en diferentes funciones celulares parece estar profundamente conservado, mientras que la “calidad” o velocidad de las enzimas individuales es lo que ha evolucionado para permitir diferentes estrategias de crecimiento.
Es como si diferentes modelos de coches (las especies bacterianas) compartieran el mismo diseño básico de motor y distribución de componentes (el plan de asignación del proteoma), pero algunos tuvieran piezas de mayor rendimiento que les permiten ir más rápido (la cinética enzimática).
Implicaciones del Descubrimiento:
- Un “Anteproyecto” Bacteriano: Sugiere la existencia de un “anteproyecto” o conjunto de reglas fundamentales y compartidas para la asignación del proteoma en bacterias muy diversas. Esto podría simplificar enormemente cómo entendemos y modelamos el comportamiento bacteriano.
- Nueva Perspectiva en Ecología Microbiana: Si diferentes bacterias comparten estos principios de asignación, podríamos empezar a pensar en ellas como “elementos químicos” con propiedades predecibles en una “tabla periódica” de la vida microbiana, facilitando la comprensión de las interacciones en comunidades complejas.
- Objetivos para la Biología Sintética: Para los ingenieros de la biología sintética que buscan optimizar bacterias para la producción de compuestos, este estudio sugiere que no solo se trata de sobreexpresar ciertos genes, sino también de entender y potencialmente modificar la cinética intrínseca de las enzimas clave.
- Preguntas Evolutivas: Plantea la intrigante pregunta de por qué no todas las bacterias han evolucionado para tener enzimas super rápidas como V. natriegens. Probablemente existan costos o compromisos asociados con esta alta velocidad que aún no comprendemos.

Conclusión: Hacia una Comprensión Más Profunda de la Vida Bacteriana
El estudio de Zhu, Mori, Hwa y Dai nos ofrece una visión refrescante y unificadora de la fisiología bacteriana. Al demostrar que la asignación del proteoma sigue un plan sorprendentemente rígido, dictado por la eficiencia ribosomal y no por la tasa de crecimiento absoluta, y que la adaptación a diferentes velocidades de crecimiento se logra principalmente a través de la modulación de la cinética enzimática, este trabajo establece una nueva base para comparar y entender el crecimiento bacteriano a través de vastas escalas filogenéticas. Es un paso crucial hacia la destilación de los principios fundamentales que gobiernan la vida en su forma más fundamental y adaptable.
Referencia del Artículo:
Zhu, M., Mori, M., Hwa, T., & Dai, X. (2025). Distantly related bacteria share a rigid proteome allocation strategy with flexible enzyme kinetics. PNAS, 122(18), e2427091122. https://doi.org/10.1073/pnas.2427091122
AI bacterias Biología Sintética CRISPR cáncer envejecimiento Escherichia coli evolución Microbioma microbiota regulación resistencia salud VIH virus
Ultimos Productos
-
Curso de Organismos Modelo en Biología Molecular Febrero 2025
El precio original era: $75.00.$57.00El precio actual es: $57.00. -
Masterclass: Dinámica y Composición de los Condensados Biomoleculares
El precio original era: $80.00.$60.00El precio actual es: $60.00.
Buscar
Últimos Posts
Últimos Comentarios
Categorías
Archivos
- marzo 2026 (5)
- febrero 2026 (15)
- enero 2026 (21)
- diciembre 2025 (20)
- noviembre 2025 (16)
- octubre 2025 (21)
- septiembre 2025 (17)
- agosto 2025 (17)
- julio 2025 (19)
- junio 2025 (24)
- mayo 2025 (26)
- abril 2025 (49)
- marzo 2025 (20)
- febrero 2025 (10)
- enero 2025 (7)
- diciembre 2024 (8)
- noviembre 2024 (23)
- octubre 2024 (7)
Palabras clave
Sígue las noticias
Te invitamos a registrar tus datos como tu correo electrónico para que puedas recibir las últimas noticias y anuncios de Biología Molecular México

By signing up, you agree to the our terms and our Privacy Policy agreement.