¡Tu carrito está actualmente vacío!
La defensa antivirus en Escherichia coli hace algo muy particular
En esta ocasión les tenemos una noticia muy particular e importante del mundo bacteriano, una defensa antiviral dependiente de una de las actividades enzimáticas más importantes, pero poco estudiadas en la biología: la ADP ribosilación [1].
La ADP ribosilación es una modificación química en proteínas muy común y que se ha encontrado en muchos seres vivos y continuamente se siguen buscando en organismos dado que tienen un papel muy importante. En el caso del humano, hay seis enzimas que hacen esta actividad biológica y participan en señalización celular, modificación estructural del genoma mediante regulación de la cromatina, regulación de la transcripción de genes, así como la reparación del ADN entre muchas otras actividades celulares. En el caso del humano, se asocia esta actividad como un mecanismo antiviral inducido por la producción de interferones, pero los blancos de esta modificación están poco estudiados. En microorganismos patógenos estas enzimas se han caracterizado como toxinas. La enzima que cataliza esta reacción se llama ADP ribosil transferasa y previamente se les conocían como PARPS por sus siglas al ser poli ADP ribosil polimerasas, es decir, son enzimas que añaden esta modificación (transferir la fracción de ribosa ADP del NAD+ a otras biomoléculas) postraduccional varias veces en una misma proteína.
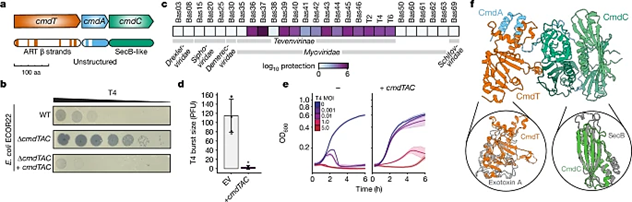
En el trabajo que deseamos compartir en esta página es del grupo de Michael T. Laub, un científico del Instituto Tecnológico de Massachussets (MIT) quien ha trabajado con nuevos sistemas que evitan los fagos (virus que infectan bacterias) puedan ejercer su replicación en bacterias y que ha encontrado nuevos sistemas de resistencia diferentes a las enzimas de restricción o el sistema CRISPR, como lo son los sistemas de toxina-antitoxina.
En este trabajo se describe una ADP-ribosil transferasa (CmdT) novedosa cuyo blanco es el ARN mensajero (mARN) haciendo que se bloquee la traducción de los genes de un fago (T4 en este reporte). Esta proteína es parte de un sistema de toxina-antitoxina que está acoplado a una chaperona (como hemos comentado en otro post de esta página, son proteínas que asisten en la conformación de proteínas en su estructura funcional nativa), y lo novedoso es que estos sistemas suelen estar asociados a controlar los sistemas de secreción. Además, CmdT modifica las adeninas de los mARNs y lo novedoso es que no tiene otros sustratos y los autores propone que quizá estas enzimas sean funcionales para modificar mARNs como parte de la inmunidad en vertebrados.

Un punto que quizá sea interesante puntualizar es que algunos mARNs en virus, bacterias y eucariontes se añaden en lugar de un cap en el extremo 5’ y que potencialmente se degradan más rápidamente [2] y que este tipo de enzimas potencialmente pueden participar en esta modificación química.
En resumen, este trabajo sugiere que la ADP ribosilación de ARNs puede ser un mecanismo mucho más amplio en la naturaleza que implique regular la traducción de genes en proteínas y que por el contrario, pueden estar involucrados tanto en mecanismos de defensa antiviral como mecanismos que están codificados en virus para aumentar su replicación. En este trabajo la proteína CmdT tiene como blanco, adeninas, pero en pares GA, lo cual se encuentra en el sitio de unión a ribosomas cuya secuencia en bacterias es AGGAGG y que en los transcritos del fago T4 son muy abundantes (GAGG). Por último, les hago la sugerencia de seguir el trabajo del profesor Laub que es muy interesante y ha aportado incluso en mecanismos evolutivos de genes [3] usando estrategias muy novedosas.
¡Felices fiestas!
Referencias
[1] Vassallo, C.N., Doering, C.R. & Laub, M.T. Anti-viral defence by an mRNA ADP-ribosyltransferase that blocks translation. Nature 636, 190–197 (2024). https://doi.org/10.1038/s41586-024-08102-8.
[2] Wiedermannová J, Babu R, Yuzenkova Y. Stochastic nature and physiological implications of 5′-NAD RNA cap in bacteria. Nucleic Acids Res. 2024 Oct 28;52(19):11838-11852. doi: 10.1093/nar/gkae813.
[3] Frumkin I, Laub MT. Selection of a de novo gene that can promote survival of Escherichia coli by modulating protein homeostasis pathways. Nat Ecol Evol. 2023 Dec;7(12):2067-2079. doi: 10.1038/s41559-023-02224-4.
AI bacterias Biología Sintética CRISPR cáncer envejecimiento Escherichia coli evolución Microbioma microbiota regulación resistencia salud VIH virus
Ultimos Productos
-
Curso de Organismos Modelo en Biología Molecular Febrero 2025
El precio original era: $75.00.$57.00El precio actual es: $57.00. -
Masterclass: Dinámica y Composición de los Condensados Biomoleculares
El precio original era: $80.00.$60.00El precio actual es: $60.00.
Buscar
Últimos Posts
Últimos Comentarios
Categorías
Archivos
- marzo 2026 (5)
- febrero 2026 (15)
- enero 2026 (21)
- diciembre 2025 (20)
- noviembre 2025 (16)
- octubre 2025 (21)
- septiembre 2025 (17)
- agosto 2025 (17)
- julio 2025 (19)
- junio 2025 (24)
- mayo 2025 (26)
- abril 2025 (49)
- marzo 2025 (20)
- febrero 2025 (10)
- enero 2025 (7)
- diciembre 2024 (8)
- noviembre 2024 (23)
- octubre 2024 (7)
Palabras clave
Sígue las noticias
Te invitamos a registrar tus datos como tu correo electrónico para que puedas recibir las últimas noticias y anuncios de Biología Molecular México

By signing up, you agree to the our terms and our Privacy Policy agreement.