¡Tu carrito está actualmente vacío!
Desactivando a Salmonella: Descubren Molécula Sintética que Bloquea su Regulador Central de Virulencia
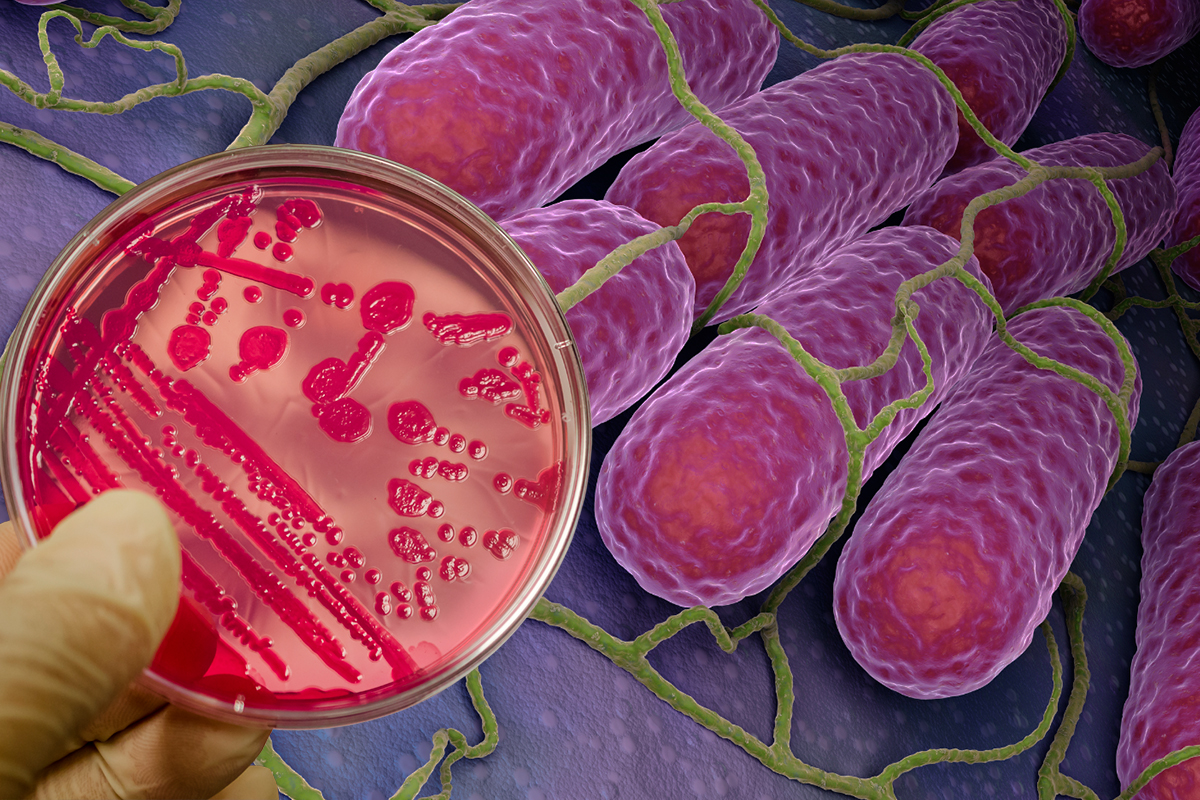
La resistencia antimicrobiana (RAM) es, sin duda, uno de los mayores desafíos para la salud global en el siglo XXI. Bacterias patógenas como Salmonella enterica, causante de cuadros que van desde gastroenteritis hasta graves infecciones sistémicas (fiebre tifoidea), están evolucionando y volviéndose insensibles a nuestros antibióticos. Ante este panorama, la comunidad científica explora estrategias alternativas, y un reciente estudio publicado en Science Advances [1] presenta un avance significativo.
Investigadores han identificado y caracterizado una molécula sintética, denominada C26, que funciona como un potente agente antivirulencia contra Salmonella.
El Arsenal de Invasión de Salmonella y su “Comandante en Jefe”
Para causar enfermedad, Salmonella necesita invadir activamente las células del hospedador. Su estrategia implica el despliegue de Sistemas de Secreción Tipo III (T3SS), estructuras moleculares complejas que actúan como nano-jeringas para inyectar proteínas efectoras, proteínas que realizan algún cambio fisiológico directamente en el citoplasma de la célula huésped. Estas proteínas manipulan la maquinaria celular, facilitando la entrada y replicación bacteriana. La producción y ensamblaje de este arsenal están codificados en regiones genéticas específicas llamadas Islas de Patogenicidad de Salmonella (SPIs).
La activación coordinada de estos genes de virulencia no es azarosa. Está orquestada por una red regulatoria compleja, cuyo “comandante en jefe” o regulador transcripcional central es la proteína HilD. Esta proteína inicia una cascada de señalización que activa la expresión de genes en múltiples SPIs (incluyendo SPI-1, esencial para el T3SS-1 de invasión, y SPI-4, para la adhesión), actuando como un interruptor maestro para la patogenicidad temprana.
De la Simulación al Laboratorio: El Hallazgo de C26
El equipo de investigación combinó enfoques computacionales y experimentales. Inicialmente, realizaron un screening virtual (cribado computacional) masivo, evaluando casi medio millón de moléculas en busca de potenciales inhibidores de componentes estructurales del T3SS-1. A partir de ahí, seleccionaron las candidatas más prometedoras para validación experimental (screening fenotípico).

Fue así como identificaron a C26, una molécula pequeña con buenas propiedades farmacológicas (“drug-like”). Sin embargo, análisis más profundos revelaron que el mecanismo de acción de C26 era más interesante de lo esperado: no actuaba sobre la estructura del T3SS, sino directamente sobre el regulador maestro, HilD.
Un Mecanismo de Inhibición Singular
Mediante análisis transcriptómicos (RNA-seq), genéticos y biofísicos (como nanoDSF y EMSA), los científicos demostraron que C26 se une a HilD, probablemente en una cavidad o pocket específico dentro de su estructura. Esta unión inhibe la capacidad de HilD para activar la expresión de sus genes diana (como hilA, el activador directo del T3SS-1).

Lo notable es que, a diferencia de otros inhibidores de HilD descritos previamente (como ciertos ácidos grasos o ácidos biliares que funcionan evitando la formación de dímeros de HilD), C26 inhibe la función de HilD sin afectar significativamente su estado de dimerización. Los datos sugieren que C26 induce un cambio conformacional o interfiere directamente con la interacción HilD-ADN, proponiendo un mecanismo de acción distinto y específico. Además, demostraron selectividad, ya que C26 no afectaba la actividad del homólogo más cercano de HilD, la proteína HilC.

Impacto Funcional y Relevancia Clínica
El efecto molecular se tradujo en consecuencias funcionales claras:
- C26 suprimió eficazmente la secreción de proteínas efectoras a través del T3SS-1 y T1SS.

- Consecuentemente, redujo drásticamente la capacidad de Salmonella para invadir células epiteliales (HeLa y MDCK) en modelos in vitro.

- También afectó la actividad del T3SS-2, importante para la supervivencia intracelular, medido en ensayos dentro de macrófagos.
- Crucialmente, C26 demostró actividad no solo contra cepas de laboratorio, sino también contra un panel diverso de aislados clínicos de S. Typhimurium (y otras serovares como S. Agona y S. Derby), independientemente de sus perfiles de resistencia a antibióticos.

- Se identificaron análogos de C26 (derivados de un análisis de relación estructura-actividad, SAR) con potencia mejorada (hasta 10 veces mayor afinidad y actividad celular), validando a C26 como un excelente scaffold químico para optimización.

Antivirulencia: Una Estrategia Contra la Resistencia
El enfoque antivirulencia, como el que representa C26, es particularmente atractivo. Al desarmar a la bacteria en lugar de matarla, se reduce la presión selectiva que impulsa la evolución de resistencia. Estos agentes podrían usarse solos para controlar infecciones o en combinación con antibióticos convencionales para aumentar su eficacia y disminuir la probabilidad de fallo terapéutico.
Conclusión y Perspectivas
El descubrimiento de C26 y la elucidación de su mecanismo de acción sobre HilD representan un avance conceptual y práctico importante. Proporciona una nueva herramienta química para estudiar la regulación de la virulencia en Salmonella y, lo más importante, ofrece un prometedor punto de partida para el desarrollo de una nueva clase de fármacos capaces de combatir infecciones bacterianas en la era de la resistencia antimicrobiana. El camino hacia la clínica es complejo, pero la identificación de dianas y moléculas innovadoras como C26 es fundamental para rearmar nuestro arsenal terapéutico.
Referencia:
[1] Boudrioua, A., Joiner, J.D., Grin, I., et al. (2025). Discovery of synthetic small molecules targeting the central regulator of Salmonella pathogenicity. Science Advances, 11, eadr5235. DOI: 10.1126/sciadv.adr5235
AI bacterias Biología Sintética CRISPR cáncer envejecimiento Escherichia coli evolución Microbioma microbiota regulación resistencia salud VIH virus
Ultimos Productos
-
Curso de Organismos Modelo en Biología Molecular Febrero 2025
El precio original era: $75.00.$57.00El precio actual es: $57.00. -
Masterclass: Dinámica y Composición de los Condensados Biomoleculares
El precio original era: $80.00.$60.00El precio actual es: $60.00.
Buscar
Últimos Posts
Últimos Comentarios
Categorías
Archivos
- marzo 2026 (5)
- febrero 2026 (15)
- enero 2026 (21)
- diciembre 2025 (20)
- noviembre 2025 (16)
- octubre 2025 (21)
- septiembre 2025 (17)
- agosto 2025 (17)
- julio 2025 (19)
- junio 2025 (24)
- mayo 2025 (26)
- abril 2025 (49)
- marzo 2025 (20)
- febrero 2025 (10)
- enero 2025 (7)
- diciembre 2024 (8)
- noviembre 2024 (23)
- octubre 2024 (7)
Palabras clave
Sígue las noticias
Te invitamos a registrar tus datos como tu correo electrónico para que puedas recibir las últimas noticias y anuncios de Biología Molecular México

By signing up, you agree to the our terms and our Privacy Policy agreement.