¡Tu carrito está actualmente vacío!
Descubriendo la estructura 3D del “segundo cerebro” al desarrollarse

El sistema nervioso entérico (ENS), a menudo apodado el “segundo cerebro” por su capacidad para funcionar de forma autónoma, es una red compleja de neuronas y células gliales incrustadas a lo largo de todo el tracto gastrointestinal. Este sistema crucial regula funciones esenciales como la motilidad intestinal, la función de barrera y el equilibrio hídrico. Comprender su desarrollo es fundamental para abordar trastornos gastrointestinales debilitantes como la enfermedad de Hirschsprung.
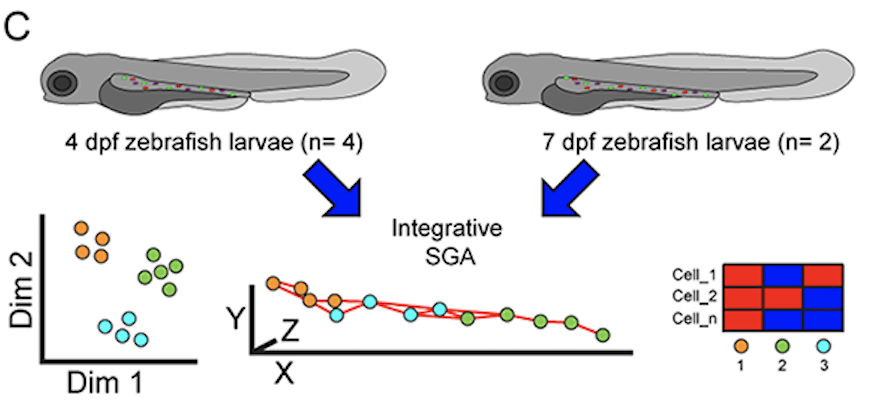
En un estudio reciente publicado como preprint en bioRxiv, investigadores han utilizado un enfoque de vanguardia, el análisis genómico espacial (SGA), para explorar la regionalización molecular del ENS en desarrollo en el pez cebra con resolución celular y espacial en 3D. El pez cebra es un modelo ideal para estos estudios debido a su alta fecundidad, conservación genética con humanos, vías moleculares compartidas y transparencia embrionaria.
El equipo desarrolló una metodología para integrar datos espaciales en 3D y transcripcionales de etapas tempranas y tardías del desarrollo del ENS en pez cebra (4 y 7 días post-fertilización). Esto permitió identificar grupos celulares regionalizados y analizar la similitud de su coexpresión génica. Mediante el análisis de 12 genes marcadores del ENS, pudieron visualizar redes en 3D del ENS intacto alrededor del intestino y predecir propiedades de conectividad celular basadas en la etapa de desarrollo.
Entre los hallazgos clave, los investigadores identificaron genes espacialmente variables, como hoxb5b, hoxa4a, etv1 y ret, que mostraron regionalización a lo largo de los ejes del intestino. Esto sugiere que estos genes podrían desempeñar un papel crucial en el control espaciotemporal preciso del desarrollo del ENS.
El estudio también reveló diferencias en la estructura de la red neuronal a lo largo del desarrollo. Se observó una mayor interconectividad y un mayor número de nodos de red en el ENS más maduro (7 dpf) en comparación con las etapas tempranas (4 dpf). Además, se identificaron genes diferencialmente expresados que definen los distintos grupos neuronales encontrados. Por ejemplo, los grupos tempranos (4 dpf) mostraron alta expresión de hoxb5b y hoxa4a, mientras que etv1 se expresó predominantemente en las etapas tardías (7 dpf). La expresión espacial de genes como nos1 y elavl3 se encontró incrementada en la región del intestino posterior en etapas tempranas y tardías, lo cual concuerda con hallazgos previos sobre la regionalización de subtipos neuronales en el pez cebra.
Interesantemente, el gen ret, conocido por su importancia en la proliferación, migración y diferenciación de progenitores neuronales entéricos (ENPs), no apareció como un gen diferencialmente expresado, pero sí como uno de los genes espacialmente variables más importantes, con un patrón hacia el frente de migración del ENS en el intestino posterior. Esto subraya la importancia de ret para la arquitectura tisular y organización espacial del ENS. Otro hallazgo fascinante fue la distribución espacial contrastante de ret y etv1 en etapas tardías, con etv1 enriquecido en el intestino anterior y medio, y ret en el intestino posterior. Esta binarización espacial sugiere patrones de diferenciación regional en 3D a lo largo del ENS del pez cebra.
La aplicación del análisis genómico espacial al desarrollo del ENS proporciona nuevas perspectivas sobre sus redes transcripcionales celulares e interacciones. Este conjunto de datos sirve como línea de base para avanzar en nuestra comprensión de los trastornos del neurodesarrollo intestinal, como la enfermedad de Hirschsprung y otras neuropatías entéricas congénitas.
Este estudio pionero, realizado por investigadores de la Universidad Rice en Houston, Texas, incluyendo al mexicano Rodrigo Moreno-Campos, resalta el poder de las técnicas de análisis espacial para desentrañar la complejidad del desarrollo del sistema nervioso.
Puedes encontrar el preprint completo aquí:
- Whole-gut spatial genomic analysis reveals molecular regionalization of the differentiating zebrafish enteric nervous system
https://x.com/uribelabrice/status/1914360949768622515
Este trabajo fue apoyado por los Institutos Nacionales de Salud (NIH) y la Fundación Nacional de Ciencias (NSF).
¿Te gustaría saber más sobre alguna parte específica de este estudio o sobre el sistema nervioso entérico?
AI bacterias Biología Sintética CRISPR cáncer envejecimiento Escherichia coli evolución Microbioma microbiota regulación resistencia salud VIH virus
Ultimos Productos
-
Curso de Organismos Modelo en Biología Molecular Febrero 2025
El precio original era: $75.00.$57.00El precio actual es: $57.00. -
Masterclass: Dinámica y Composición de los Condensados Biomoleculares
El precio original era: $80.00.$60.00El precio actual es: $60.00.
Buscar
Últimos Posts
Últimos Comentarios
Categorías
Archivos
- marzo 2026 (5)
- febrero 2026 (15)
- enero 2026 (21)
- diciembre 2025 (20)
- noviembre 2025 (16)
- octubre 2025 (21)
- septiembre 2025 (17)
- agosto 2025 (17)
- julio 2025 (19)
- junio 2025 (24)
- mayo 2025 (26)
- abril 2025 (49)
- marzo 2025 (20)
- febrero 2025 (10)
- enero 2025 (7)
- diciembre 2024 (8)
- noviembre 2024 (23)
- octubre 2024 (7)
Palabras clave
Sígue las noticias
Te invitamos a registrar tus datos como tu correo electrónico para que puedas recibir las últimas noticias y anuncios de Biología Molecular México

By signing up, you agree to the our terms and our Privacy Policy agreement.