¡Tu carrito está actualmente vacío!
Determinantes Genómicos de la Jerarquía de Expresión Antigénica en Tripanosomas Africanos
La variación antigénica es una estrategia crucial de evasión inmune empleada por diversos patógenos, incluido Trypanosoma brucei, el agente causante de la tripanosomiasis africana humana (HAT por sus siglas en inglés) y la enfermedad del sueño en animales. Este parásito cambia periódicamente su antígeno de superficie, la glicoproteína variable de superficie (o VSGs), lo que le permite evadir la respuesta inmune del hospedero. Comprender los mecanismos genómicos que impulsan esta variación antigénica es vital para entender la patogénesis del tripanosoma y desarrollar posibles intervenciones moleculares para evitar o reducir el impacto en la salud de los humanos por este parásito. Este estudio de Keneskhanova y colaboradores [1] investiga los determinantes genómicos que influyen en la jerarquía de expresión antigénica en T. brucei utilizando secuenciación de ARN de célula única o single cell RNAseq (scRNA-seq).
Antecedentes
El genoma de T. brucei contiene aproximadamente 2,500 genes que codifican para VSGs, en su mayoría silenciosos, con solo uno expresado a la vez desde un sitio de expresión telomérico, cuando el parásito se encuentra en el torrente sanguíneo. El cambio antigénico puede ocurrir a través de modificaciones transcripcionales o eventos de recombinación homóloga, generalmente desencadenados por rupturas de doble cadena de ADN. Estudios previos sugieren que la variación antigénica sigue un patrón jerárquico, pero los determinantes genómicos subyacentes siguen siendo poco conocidos. Mediante un enfoque novedoso de scRNA-seq, este estudio busca esclarecer los mecanismos moleculares que regulan el cambio de VSGs.
Métodos
Enfoque de Secuenciación de scRNA-seq
Los investigadores desarrollaron un protocolo adaptado para tripanosomas basado en Smart-seq3xpress, denominado SL-Smart-seq3xpress. Este método mejora la sensibilidad y especificidad en la detección de transcritos de VSG, permitiendo el seguimiento en tiempo real de los eventos de cambio antigénico a nivel de células individuales. En comparación con métodos anteriores, este enfoque proporciona una mayor resolución en el análisis de cambios en la expresión génica y eventos de recombinación de ADN.

Inducción de Rupturas de Doble Cadena con CRISPR-Cas9
Para investigar cómo las rupturas de doble cadena en el ADN influyen en el cambio de VSGs, los autores usaron el sistema de CRISPR-Cas9 para introducir rupturas de doble cadena dirigidas en ubicaciones específicas dentro del gen VSG activo. Analizando los resultados de estas rupturas, los autores pretendían determinar el papel de los moldes usados durante la recombinación homóloga en la selección del nuevo gen codificante para VSG que será expresado.
Resultados
Las Rupturas de Doble Cadena Inducen el Cambio de VSG
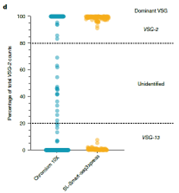
Tras inducir una ruptura de doble cadena en el gen VSG activamente expresado, más del 94% de la población parasitaria perdió la expresión del gen activo en un lapso de cuatro días. El análisis transcriptómico de célula única reveló que las células activaron únicamente VSGs adyacentes al telómero. Esto sugiere que la ubicación genómica juega un papel crítico en la selección de los VSGs que serán expresados tras un evento de ruptura de doble cadena.
La Vía de Reparación Determina el Resultado de Expresión Antigénica
Se identificaron dos mecanismos principales de reparación:
- Conversión Génica Segmentaria: Si existía una secuencia VSG homóloga disponible, el parásito utilizaba la conversión génica para reparar la ruptura de doble cadena, incorporando material genético de genes VSG silenciosos o pseudogenes. Esto resultaba en la formación de nuevos genes VSG en mosaico, aumentando la diversidad antigénica.
- Replicación Inducida por Ruptura (BIR): En los casos donde no había una plantilla de reparación homóloga disponible, el parásito activaba un VSG adyacente al telómero mediante BIR. Este mecanismo se observó predominantemente en células que expresaban un gen VSG especifico y activo (VSG-2), ya que este gen tiene poca homología con otros genes VSG.
Selección Jerárquica de Antígenos
El estudio demostró que la expresión antigénica sigue un patrón semi predecible influenciado por la disponibilidad de secuencias de reparación homólogas. Las células activaban preferentemente VSGs con mayor similitud genómica con el gen dañado. Además, se observó que el cambio transcripcional a un gen codificante diferente ocurrió inicialmente, pero con el tiempo fue superado por células que reactivaban el gen codificante para VSG-2 original, lo que sugiere una ventaja reproductiva en la transcripción de este gen o el primer gen que expresa la población de parásitos.
Discusión
Restricciones Genómicas en la Variación Antigénica
Los hallazgos revelan que la jerarquía de expresión de VSG está determinada por la organización genómica de los genes VSG y los mecanismos de reparación de rupturas de doble cadena. La presencia de un molde de reparación homóloga determina si el cambio antigénico ocurre mediante conversión génica segmentaria o por BIR.

Implicaciones para la Evasión Inmune del Tripanosoma
La preferencia por la conversión génica segmentaria cuando hay plantillas homólogas disponibles sugiere que los tripanosomas pueden priorizar esta vía para aumentar la diversidad antigénica. Este proceso permite al parásito generar variantes antigénicas novedosas sin interrumpir el gen activo, asegurando así una estrategia continua y efectiva de evasión inmune.
Direcciones Futuras
Se requiere más investigación para explorar cómo se regulan estas vías de reparación a nivel molecular. Comprender los mecanismos que gobiernan la búsqueda de homología y la selección de plantillas de reparación podría proporcionar información clave para el desarrollo de posibles tratamientos contra las infecciones por T. brucei.
Conclusión
Este estudio resalta el papel crucial de los mecanismos de reparación del ADN en la configuración de la variación antigénica (por tanto, la evasión del sistema inmune) en T. brucei. Los hallazgos enfatizan que la selección antigénica no es completamente aleatoria, sino que está influenciada por la arquitectura genómica y la disponibilidad de vías de reparación. Mediante el uso de técnicas avanzadas de transcriptómica de célula única, este trabajo proporciona nuevas perspectivas sobre los fundamentos moleculares de la variación antigénica y abre camino para futuras investigaciones sobre las estrategias de evasión inmune del tripanosoma.
Referencias
Keneskhanova Z, McWilliam KR, Cosentino RO, Barcons-Simon A, Dobrynin A, Smith JE, Subota I, Mugnier MR, Colomé-Tatché M, Siegel TN. Genomic determinants of antigen expression hierarchy in African trypanosomes. Nature. 2025 Mar 12. doi: 10.1038/s41586-025-08720-w.
AI bacterias Biología Sintética CRISPR cáncer envejecimiento Escherichia coli evolución Microbioma microbiota regulación resistencia salud VIH virus
Ultimos Productos
-
Curso de Organismos Modelo en Biología Molecular Febrero 2025
El precio original era: $75.00.$57.00El precio actual es: $57.00. -
Masterclass: Dinámica y Composición de los Condensados Biomoleculares
El precio original era: $80.00.$60.00El precio actual es: $60.00.
Buscar
Últimos Posts
Últimos Comentarios
Categorías
Archivos
- marzo 2026 (5)
- febrero 2026 (15)
- enero 2026 (21)
- diciembre 2025 (20)
- noviembre 2025 (16)
- octubre 2025 (21)
- septiembre 2025 (17)
- agosto 2025 (17)
- julio 2025 (19)
- junio 2025 (24)
- mayo 2025 (26)
- abril 2025 (49)
- marzo 2025 (20)
- febrero 2025 (10)
- enero 2025 (7)
- diciembre 2024 (8)
- noviembre 2024 (23)
- octubre 2024 (7)
Palabras clave
Sígue las noticias
Te invitamos a registrar tus datos como tu correo electrónico para que puedas recibir las últimas noticias y anuncios de Biología Molecular México

By signing up, you agree to the our terms and our Privacy Policy agreement.