¡Tu carrito está actualmente vacío!
Mapeando al Enemigo Microscópico: Crean un Atlas Genético Sin Precedentes del Hongo Mortal Cryptococcus neoformans
En Pocas Palabras:
Cryptococcus neoformans es un hongo que causa meningitis fúngica, especialmente en personas con sistemas inmunes debilitados, y es una de las principales prioridades de la OMS. Para entender mejor cómo este patógeno causa enfermedad y cómo podríamos combatirlo, un equipo de científicos construyó y analizó una colección masiva de más de 4,300 cepas mutantes de este hongo, cada una con un gen diferente inactivado. Estudiaron cómo crecía cada mutante bajo 141 condiciones de laboratorio diferentes y durante una infección en ratones. Este “mapa fenotípico” de alta resolución reveló más de 100 “módulos funcionales” (grupos de genes que trabajan juntos) y descubrió que Cryptococcus utiliza maquinaria celular similar a la de los animales (que se creía exclusiva de ellos) para sentir su entorno y reparar su ADN. También identificó nuevos factores cruciales para su virulencia, incluyendo mecanismos de adaptación ambiental que el hongo “reutiliza” para infectar mamíferos. Este recurso sin precedentes abre nuevas vías para desarrollar terapias antifúngicas urgentemente necesarias.

Descifrando a un Asesino Fúngico Prioritario
Las infecciones fúngicas representan una amenaza creciente y a menudo subestimada para la salud global, causando millones de muertes cada año, especialmente en individuos inmunocomprometidos. En la cima de la lista de patógenos fúngicos prioritarios de la Organización Mundial de la Salud (OMS) se encuentra Cryptococcus neoformans, un hongo ambiental que, tras ser inhalado, puede diseminarse al cerebro y causar una meningitis fúngica devastadora, responsable de casi el 20% de las muertes relacionadas con el SIDA. A pesar de su importancia clínica, nuestra comprensión de la biología fundamental de este patógeno y de los mecanismos que le permiten infectar a los mamíferos es incompleta. Un monumental estudio publicado en la prestigiosa revista Cell por Michael J. Boucher, Hiten D. Madhani y un extenso equipo de colaboradores de la Universidad de California en San Francisco y otras instituciones, ha dado un paso de gigante para llenar este vacío. Han construido y analizado fenotípicamente la mayor colección de mutantes de deleción génica de C. neoformans hasta la fecha, creando un mapa funcional de alta resolución de su genoma que revela su biología única y los motores moleculares de su patogenicidad.
Un Hongo Único con Secretos por Descubrir
C. neoformans es un basidiomiceto, un linaje fúngico que divergió de los hongos modelo más estudiados (como las levaduras de panadería Saccharomyces cerevisiae o Schizosaccharomyces pombe, que son ascomicetos) hace más de 450 millones de años. Esto significa que casi la mitad de sus genes no tienen equivalentes directos (ortólogos) en estas levaduras modelo, y muchos son únicos de los basidiomicetos. Paradójicamente, C. neoformans ha conservado algunas maquinarias celulares complejas, como el silenciamiento génico y el splicing de ARN, que se han perdido o simplificado en las levaduras modelo, convirtiéndolo en un sistema experimental valioso también para estudiar biología eucariota fundamental.
La Hazaña: Más de 4,300 Mutantes Bajo la Lupa
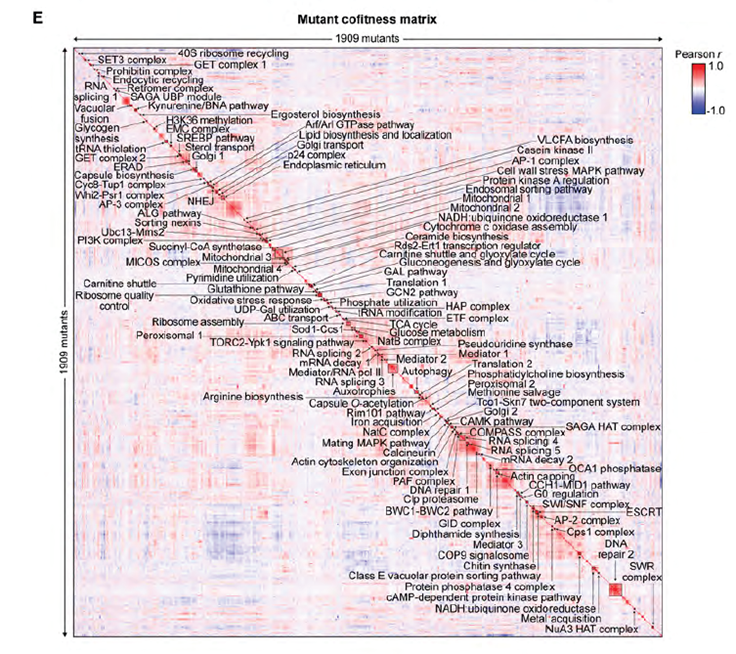
Para descifrar globalmente la función de los genes de C. neoformans, los investigadores se embarcaron en un proyecto ambicioso: construir una colección de cepas donde cada gen (o casi cada gen no esencial) fuera sistemáticamente eliminado (noqueado o “knockout”). Lograron generar 4,328 cepas de deleción génica validadas, cubriendo aproximadamente el 79% de los genes no esenciales predichos.

Luego, sometieron esta vasta biblioteca de mutantes a un escrutinio exhaustivo, utilizando una técnica de secuenciación de alta precisión desarrollada por ellos mismos llamada KO-seq. Esta técnica permite medir la “aptitud” (fitness) o capacidad de crecimiento de cada mutante de forma masiva y en paralelo cuando se cultivan todos juntos en un mismo “pool”. Cada mutante fue analizado en:
- 141 condiciones de crecimiento in vitro diversas: variando medios de cultivo, temperaturas, pH, fuentes de carbono, estresores químicos y fármacos antifúngicos.
- Un modelo de infección en ratones: para identificar los genes necesarios para causar enfermedad (virulencia).

Un Atlas Fenotípico Revela la Lógica Interna del Hongo
El resultado de estos millones de mediciones de fitness es un “paisaje fenotípico” increíblemente detallado, un atlas que conecta genes con funciones bajo diferentes contextos. Al agrupar genes con perfiles fenotípicos similares (es decir, genes cuyos mutantes se comportan de manera similar en las diferentes condiciones), los investigadores identificaron más de 100 “módulos funcionales”, grupos de genes que probablemente trabajan juntos en una misma vía o proceso celular.
Este atlas permitió hacer descubrimientos notables:
- Maquinaria Celular “Animal” en un Hongo:
- Sensor de pH tipo Hedgehog: Descubrieron que la vía Rim101, crucial para que el hongo sienta y se adapte al pH alcalino (como el que encontraría en el huésped), utiliza componentes (Rra1, Rra2) que forman un complejo sorprendentemente similar al sensor de pH de la vía Hedgehog en animales, un mecanismo que se creía específico de metazoos.
- Vía de Reparación de ADN tipo Shieldina/53BP1: Identificaron una nueva proteína, Njf1, que junto con otras (Rev7, Rif1), forma un complejo análogo al sistema Shieldina/53BP1 de los animales, el cual es crucial para decidir cómo reparar las roturas del ADN de doble cadena (favoreciendo la unión de extremos no homólogos, NHEJ). Esta vía se había perdido en las levaduras modelo.
- Homólogos del Canal de Sodio NALCN (UNC79/UNC80): Encontraron que C. neoformans posee ortólogos de las proteínas animales UNC79 y UNC80, que son componentes de un canal de sodio crucial para la excitabilidad neuronal. En el hongo, estas proteínas parecen estar involucradas en la activación de la vía de la calcineurina (importante para la tolerancia al estrés y la virulencia).


- Motores de Virulencia y Adaptación al Huésped:
- Complejo de Síntesis de Ácido Hialurónico con Tetraspanina: Identificaron un complejo que incluye la sintasa de ácido hialurónico (Cps1, un conocido factor de virulencia) y una proteína tetraspanina (Tsp2). Las tetraspaninas son importantes en la organización de la membrana en animales, pero su papel en hongos era menos claro. Demostraron que este complejo es esencial para la producción de la cápsula de ácido hialurónico y la neurovirulencia (capacidad de invadir el cerebro).
- Factores Secretados y Glucanos de Pared: Identificaron proteínas secretadas (como una nueva oxidasa de glioxal llamada Glx1) y genes implicados en la modificación de la pared celular (como Rgh1, que parece regular la producción de α-glucano en respuesta a condiciones similares a las del huésped) que son cruciales para la infección. Curiosamente, parece que C. neoformans ha “reutilizado” mecanismos de adaptación ambiental para promover su virulencia en mamíferos, a pesar de no tener un reservorio animal conocido.
Implicaciones y Futuro: Un Recurso de Valor Incalculable
Este estudio representa un hito en la micología y la investigación de patógenos:
- Recurso Comunitario: La colección de mutantes y el vasto conjunto de datos fenotípicos son un recurso sin precedentes que estará disponible para toda la comunidad científica, acelerando la investigación sobre C. neoformans y otros hongos.
- Nuevas Dianas Terapéuticas: La identificación de genes y vías esenciales para la virulencia o la supervivencia bajo estrés específico del huésped abre la puerta al desarrollo de nuevos fármacos antifúngicos, que se necesitan con urgencia.
- Comprensión de la Biología Fúngica y Eucariota: Los hallazgos sobre maquinaria celular conservada (como los sensores de pH o las vías de reparación de ADN) proporcionan nuevas perspectivas sobre la evolución de procesos eucariotas fundamentales.
- Modelo para Otros Patógenos: La metodología KO-seq y el enfoque de fenotipado masivo pueden aplicarse a otros patógenos difíciles de estudiar, acelerando nuestra comprensión de cómo causan enfermedades.
Conclusión: Iluminando la Oscuridad de un Patógeno Mortal
El trabajo de Boucher, Madhani y su equipo es una demostración magistral de cómo la genómica funcional a gran escala puede transformar nuestra comprensión de un patógeno complejo y peligroso. Al construir y analizar meticulosamente miles de mutantes de Cryptococcus neoformans, han creado un atlas que no solo revela la biología única de este hongo, sino que también identifica sus puntos débiles y los mecanismos que lo convierten en un formidable invasor. Este estudio no solo es una proeza técnica, sino una fuente de conocimiento que, sin duda, impulsará la búsqueda de nuevas estrategias para combatir las devastadoras infecciones fúngicas que amenazan la salud global.
Referencia del Artículo:
Boucher, M. J., Banerjee, S., Joshi, M. B., Wei, A. L., Nalley, M. J., Huang, M. Y., … & Madhani, H. D. (2025). Phenotypic landscape of an invasive fungal pathogen reveals its unique biology. Cell, 188, 1–22. https://doi.org/10.1016/j.cell.2025.05.017
(Fecha de publicación prevista: 24 de julio de 2025).
AI bacterias Biología Sintética CRISPR cáncer envejecimiento Escherichia coli evolución Microbioma microbiota regulación resistencia salud VIH virus
Ultimos Productos
-
Curso de Organismos Modelo en Biología Molecular Febrero 2025
El precio original era: $75.00.$57.00El precio actual es: $57.00. -
Masterclass: Dinámica y Composición de los Condensados Biomoleculares
El precio original era: $80.00.$60.00El precio actual es: $60.00.
Buscar
Últimos Posts
Últimos Comentarios
Categorías
Archivos
- marzo 2026 (5)
- febrero 2026 (15)
- enero 2026 (21)
- diciembre 2025 (20)
- noviembre 2025 (16)
- octubre 2025 (21)
- septiembre 2025 (17)
- agosto 2025 (17)
- julio 2025 (19)
- junio 2025 (24)
- mayo 2025 (26)
- abril 2025 (49)
- marzo 2025 (20)
- febrero 2025 (10)
- enero 2025 (7)
- diciembre 2024 (8)
- noviembre 2024 (23)
- octubre 2024 (7)
Palabras clave
Sígue las noticias
Te invitamos a registrar tus datos como tu correo electrónico para que puedas recibir las últimas noticias y anuncios de Biología Molecular México

By signing up, you agree to the our terms and our Privacy Policy agreement.