¡Tu carrito está actualmente vacío!
Proteínas que Computan: Redes de Dimerización Revelan una Sofisticada Lógica Celular Específica de Contexto
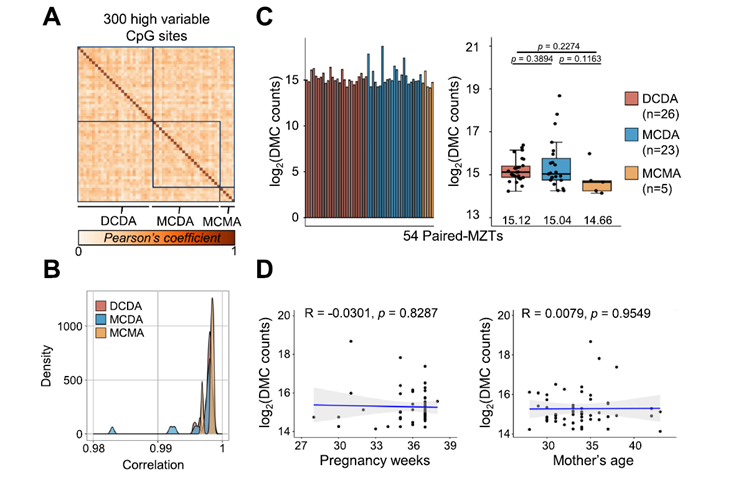
Investigadores del Caltech y colaboradores [1] han revelado cómo redes de proteínas que compiten para formar dímeros (parejas) pueden realizar cálculos bioquímicos complejos, adaptando sus respuestas a diferentes contextos celulares, según publican en la revista Cell. Este trabajo proporciona una nueva perspectiva sobre cómo las células procesan información utilizando un conjunto limitado de componentes moleculares.
Muchas vías de señalización biológica dependen de familias de proteínas (monómeros) que se unen entre sí para formar dímeros. Estos dímeros a menudo son las unidades funcionales activas, por ejemplo, uniéndose al ADN para regular la expresión génica. Sin embargo, en muchas familias de proteínas, la formación de dímeros es competitiva y “muchos a muchos”: un monómero puede potencialmente formar pareja con varios otros monómeros diferentes.
El equipo de investigación, liderado por Michael B. Elowitz, utilizó un enfoque computacional sistemático para explorar las capacidades de estas redes de dimerización competitiva. Modelaron estas redes considerando las concentraciones de monómeros como entradas (inputs) y las concentraciones de dímeros específicos como salidas (outputs).

Hallazgos Clave:
- Computación Diversa: Descubrieron que incluso redes pequeñas (con solo 3 a 6 tipos de monómeros) son sorprendentemente “expresivas”, capaces de realizar una amplia gama de funciones de procesamiento de señales, incluyendo respuestas no monótonas (como funciones “bump”, donde la salida es alta solo a niveles intermedios de entrada) y funciones lógicas multi entrada (como puertas AND, NAND, XOR, NIMPLY).
- Versatilidad y Contextualidad: Un hallazgo crucial es la “versatilidad” de estas redes. Una misma red de proteínas (con las mismas afinidades de interacción) puede realizar diferentes cálculos simplemente cambiando los niveles de expresión de sus componentes monoméricos “accesorios” (aquellos que no son la señal de entrada directa). Esto explica cómo diferentes tipos celulares, que expresan los mismos tipos de proteínas, pero a diferentes niveles, pueden interpretar las mismas señales de entrada de maneras distintas, llevando a respuestas específicas del tipo celular.
- Escalabilidad con Tamaño y Conectividad: La capacidad computacional (expresividad) aumenta con el tamaño de la red (número de monómeros) y su conectividad (número de interacciones posibles), aunque con rendimientos decrecientes más allá de cierto tamaño para funciones básicas. Funciones más complejas requieren redes más grandes.
- Poder de las Redes Aleatorias: Sorprendentemente, el estudio muestra que redes grandes con afinidades de interacción aleatorias (como las que podrían surgir por duplicación y divergencia evolutiva) pueden ser ajustadas para realizar casi todas las funciones computacionales posibles (para una entrada) simplemente sintonizando los niveles de expresión de los monómeros accesorios. Esto sugiere que la evolución no necesita esculpir con precisión todas las afinidades de interacción; ajustar los niveles de expresión puede ser una estrategia muy poderosa y flexible.
- Relevancia Biológica: Las redes de dimerización naturales, como las familias de factores de transcripción bZIP, receptores nucleares (NR) y proteínas bHLH, a menudo tienen tamaños y patrones de expresión co-expresados en tipos celulares que, según el modelo, son suficientes para realizar estas computaciones complejas y específicas de contexto. El modelo también funciona en escalas de tiempo (minutos a horas) y con niveles de robustez al ruido relevantes biológicamente.
Importancia del Estudio:
Este trabajo establece un marco fundamental para entender las redes de dimerización competitiva como sistemas computacionales. Explica cómo la simple competencia estequiométrica puede generar comportamientos complejos y adaptativos. Proporciona una explicación plausible para la prevalencia de estas redes en la biología y para cómo las células logran especificidad de respuesta en diferentes contextos utilizando un conjunto compartido de proteínas. Además, los hallazgos tienen implicaciones para la biología sintética, sugiriendo que estas redes podrían ser diseñadas para crear sensores y circuitos lógicos complejos y adaptables en células en las que se puede hacer ingeniería.

Referencia:
[1] Parres-Gold et al., Contextual computation by competitive protein dimerization networks, Cell 188, 1984–2002 (April 3, 2025). https://doi.org/10.1016/j.cell.2025.01.036
AI bacterias Biología Sintética CRISPR cáncer envejecimiento Escherichia coli evolución Microbioma microbiota regulación resistencia salud VIH virus
Ultimos Productos
-
Curso de Organismos Modelo en Biología Molecular Febrero 2025
El precio original era: $75.00.$57.00El precio actual es: $57.00. -
Masterclass: Dinámica y Composición de los Condensados Biomoleculares
El precio original era: $80.00.$60.00El precio actual es: $60.00.
Buscar
Últimos Posts
Últimos Comentarios
Categorías
Archivos
- marzo 2026 (5)
- febrero 2026 (15)
- enero 2026 (21)
- diciembre 2025 (20)
- noviembre 2025 (16)
- octubre 2025 (21)
- septiembre 2025 (17)
- agosto 2025 (17)
- julio 2025 (19)
- junio 2025 (24)
- mayo 2025 (26)
- abril 2025 (49)
- marzo 2025 (20)
- febrero 2025 (10)
- enero 2025 (7)
- diciembre 2024 (8)
- noviembre 2024 (23)
- octubre 2024 (7)
Palabras clave
Sígue las noticias
Te invitamos a registrar tus datos como tu correo electrónico para que puedas recibir las últimas noticias y anuncios de Biología Molecular México

By signing up, you agree to the our terms and our Privacy Policy agreement.