¡Tu carrito está actualmente vacío!
¿Qué vamos a hacer con la resistencia a los antibióticos?
Cada que vemos una nueva película de alguna epidemia nos entra la pregunta ¿puede pasar? COVID-19 fue un evento que muestra que las enfermedades infecciosas son cosa seria.
Ahora, la preocupación más cercana es el incremento de microorganismos resistentes a los antimicrobianos disponibles para combatir infecciones microbianas, y una pandemia causada por una bacteria multirresistente es un escenario complicado de abordar.
Primero que nada, el aislar un nuevo antibiótico y demostrar que es seguro para su uso y efectivo para eliminar microorganismos de relevancia médica (para una lista completa de bacterias de alta prioridad para la Organización Mundial de la Salud, consultar 1) no es tarea fácil. Son pocas las empresas farmacéuticas y centros de investigación desarrollando programas de descubrimiento de nuevos fármacos.
En el 2015, un grupo encontró un nuevo fármaco (el teixobactin) que parecía no generar resistencia y ser sumamente efectivo contra Staphyloccocus aureus multirresistente [2]. Este es un trabajo muy novedoso porque lograron aislar microorganismos que previamente no habían sido aislados en el laboratorio usando una técnica llamada isolation chip (o iChip por aquello de la tendencia moderna) y que permite aislar en condiciones ambientales en las que el microorganismo prolifera, ya que es un dispositivo (se muestra en la imagen siguiente), que puede permitir la difusión de nutrientes del sitio de aislamiento, pero quedan atrapados los microorganismos dentro del dispositivo y pueden ser “domesticados” para crecer en medios comunes de laboratorio.

El trabajo de Ling y colaboradores [2] parecía ser una fuente de esperanza para el tratamiento de enfermedades causadas por microorganismos multirresistentes.
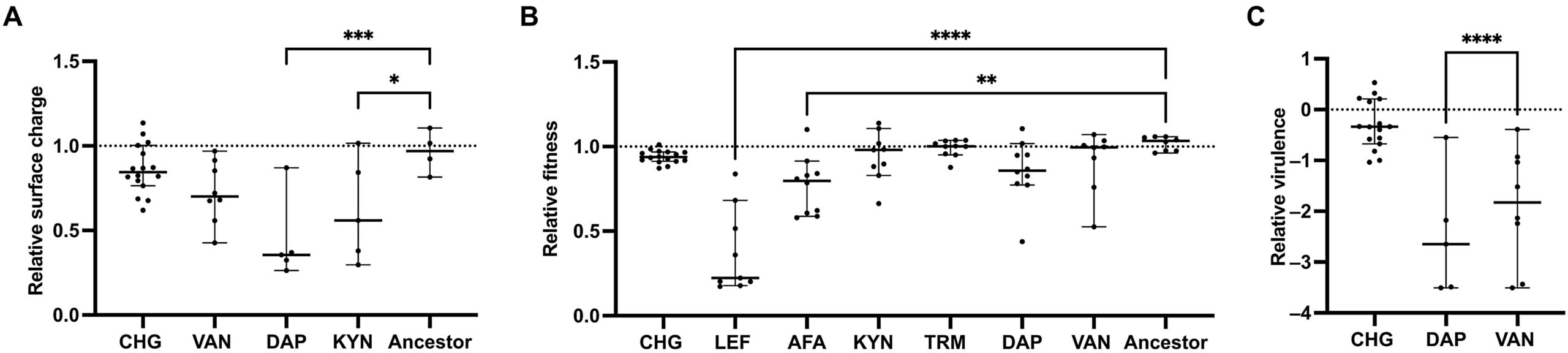
Pero ahora tenemos un nuevo problema. Recién publicado en Science Translational Medicine, Martins y colaboradores [3] reportan un trabajo muy importante en el área de resistencia a fármacos en bacterias. Haciendo uso de experimentos de evolución en el laboratorio (crecimiento continuo de microorganismos en presencia de una presión selectiva) en la que demuestran que muchos antibióticos en fase de prueba con mucha eficiencia, incluyendo el teixobactin, inducen resistencia cruzada a otros antibióticos de uso clínico como son la vancomicina, daptomicina e incluso al teixobactin que como citamos acá, es uno de los fármacos más prometedores que se han aislado. Lo más sorprendente de este estudio es que la resistencia surge de genes ya existentes en bacterias y solamente se encontraron dos genes que llegaron a acumular hasta 12 mutaciones, el resto presentaron un par de mutaciones nada más y fue suficiente para generar líneas resistentes.

El resultado de este estudio es que dentro de los nuevos fármacos que generan resistencia cruzada, hay una buna noticia: el proceso es unidireccional, es decir, producen resistencia a fármacos en uso, pero no a los nuevos fármacos que son altamente efectivos y que además abre la posibilidad de hacer modificaciones químicas en fármacos novedosos para incrementar su efectividad. Este trabajo es un nuevo esquema de trabajo para evaluar la evolución de la resistencia a fármacos y su impacto incluso en la virulencia de los microorganismos que son expuestos a estas moléculas. Además, abre una ventana que no se había evaluado antes, cómo evolucionan los microorganismos expuestos a antibióticos novedosos. Como nota adicional, este trabajo hace evidente la importancia de llevar a buen término los tratamientos con antibióticos ya que esto es la fuente principal de resistencia.
Referencias
[1] https://www.who.int/publications/i/item/9789240093461
[2] Ling LL, Schneider T, Peoples AJ, Spoering AL, Engels I, Conlon BP, Mueller A, Schäberle TF, Hughes DE, Epstein S, Jones M, Lazarides L, Steadman VA, Cohen DR, Felix CR, Fetterman KA, Millett WP, Nitti AG, Zullo AM, Chen C, Lewis K. A new antibiotic kills pathogens without detectable resistance. Nature. 2015 Jan 22;517(7535):455-9. doi: 10.1038/nature14098.
[3]Ana Martins et al. ,Antibiotic candidates for Gram-positive bacterial infections induce multidrug resistance.Sci. Transl. Med.17,eadl2103(2025).DOI:10.1126/scitranslmed.adl2103
AI bacterias Biología Sintética CRISPR cáncer envejecimiento Escherichia coli evolución Microbioma microbiota regulación resistencia salud VIH virus
Ultimos Productos
-
Curso de Organismos Modelo en Biología Molecular Febrero 2025
El precio original era: $75.00.$57.00El precio actual es: $57.00. -
Masterclass: Dinámica y Composición de los Condensados Biomoleculares
El precio original era: $80.00.$60.00El precio actual es: $60.00.
Buscar
Últimos Posts
Últimos Comentarios
Categorías
Archivos
- marzo 2026 (5)
- febrero 2026 (15)
- enero 2026 (21)
- diciembre 2025 (20)
- noviembre 2025 (16)
- octubre 2025 (21)
- septiembre 2025 (17)
- agosto 2025 (17)
- julio 2025 (19)
- junio 2025 (24)
- mayo 2025 (26)
- abril 2025 (49)
- marzo 2025 (20)
- febrero 2025 (10)
- enero 2025 (7)
- diciembre 2024 (8)
- noviembre 2024 (23)
- octubre 2024 (7)
Palabras clave
Sígue las noticias
Te invitamos a registrar tus datos como tu correo electrónico para que puedas recibir las últimas noticias y anuncios de Biología Molecular México

By signing up, you agree to the our terms and our Privacy Policy agreement.