¡Tu carrito está actualmente vacío!
ROADIES al Rescate: La Herramienta que Simplifica la Creación de Árboles Evolutivos Directamente desde Genomas Crudos
El Desafío de Trazar el Árbol de la Vida en la Era Genómica
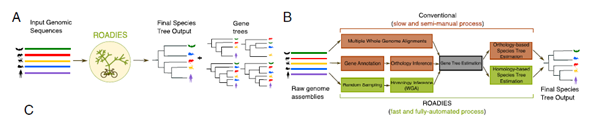
En la era de la secuenciación masiva, tenemos acceso a una cantidad sin precedentes de genomas completos de una vasta diversidad de especies. Estos datos genómicos son una mina de oro para entender las relaciones evolutivas entre los organismos, es decir, para construir “árboles de la vida” o filogenias. Sin embargo, a pesar de la facilidad para obtener los genomas, el proceso de inferir árboles filogenéticos precisos sigue siendo un cuello de botella considerable, a menudo requiriendo una profunda experiencia en bioinformática, un considerable poder computacional y flujos de trabajo complejos y poco automatizados. Para derribar estas barreras, Anshu Gupta, Siavash Mirarab y Yatish Turakhia han desarrollado ROADIES, una herramienta revolucionaria presentada en la revista PNAS. ROADIES es un pipeline completamente automatizado, escalable y fácil de usar que permite inferir árboles de especies directamente a partir de ensamblajes genómicos crudos, democratizando el acceso a análisis filogenómicos robustos.

El Problema Tradicional: Un Camino Lleno de Obstáculos
La inferencia filogenética moderna, especialmente a gran escala, enfrenta varios desafíos:
- Anotación Genómica: Identificar genes funcionales y otras regiones relevantes en genomas recién secuenciados es un proceso lento y que requiere conocimiento experto.
- Ortología: Distinguir entre ortólogos (genes que divergen por especiación) y parálogos (genes que divergen por duplicación dentro de un mismo genoma) es crucial pero complejo. Muchos métodos tradicionales se basan en encontrar conjuntos de genes de copia única.
- Alineamiento de Genomas Completos (m-WGA): Alinear múltiples genomas completos es computacionalmente muy intensivo y sensible a la calidad de un árbol guía inicial.
- Discordancia entre Genes: Diferentes genes pueden contar historias evolutivas ligeramente distintas debido a procesos como la clasificación incompleta de linajes. Los métodos modernos deben ser capaces de manejar esta “discordancia”.
- Intensidad Computacional y Experiencia: Los flujos de trabajo suelen implicar múltiples herramientas, cada una con sus propios parámetros, lo que exige mucho tiempo y conocimientos especializados.

ROADIES: Una Nueva Ruta Más Directa y Eficiente
ROADIES (cuyas siglas en inglés significan “Estimación de Árboles de Especies Consciente de la Discordancia, Libre de Referencia, Libre de Ortología y Libre de Anotación”) aborda estos problemas con un enfoque innovador:
- Sin Anotaciones, Sin Referencias, Sin Ortología Predefinida:
- Muestreo Aleatorio de Loci: En lugar de depender de genes predefinidos o anotados, ROADIES muestrea aleatoriamente segmentos cortos (llamados “genes” o c-genes, que idealmente no tienen recombinación interna) directamente de los genomas ensamblados. Esto elimina la necesidad de anotaciones previas, la dependencia de un genoma de referencia (evitando sesgos) y la engorrosa tarea de identificar ortólogos estrictos.
- Acepta Genes Multicopia: Crucialmente, ROADIES utiliza métodos (como ASTRAL-Pro3) que pueden inferir el árbol de especies a partir de árboles de genes que contienen múltiples copias por especie (parálogos), dejando que el propio método estadístico desenrede la historia evolutiva.
- Automatización Completa: Es un pipeline “de principio a fin”. El usuario proporciona los ensamblajes genómicos crudos y ROADIES se encarga de todo el proceso:
- Muestreo de loci homólogos.
- Alineamiento múltiple de secuencias para cada locus.
- Inferencia de árboles de genes individuales.
- Combinación de los árboles de genes en un árbol de especies final, teniendo en cuenta la discordancia.
- Iterativo y Convergente: ROADIES funciona de forma iterativa. Comienza con un número pequeño de loci muestreados y va aumentando este número en cada iteración hasta que el árbol de especies resultante se estabiliza y alcanza un nivel de confianza predefinido por el usuario. Esto asegura que se utiliza la cantidad adecuada de datos sin un esfuerzo computacional excesivo.
- Escalabilidad y Flexibilidad: Está diseñado para ser altamente paralelizable y escalable a cientos o miles de genomas. Ofrece diferentes modos de operación (preciso, balanceado, rápido) para adaptarse a las necesidades del usuario y los recursos disponibles.
Validación Rigurosa: ROADIES se Mide con los Grandes
Los autores probaron ROADIES en conjuntos de datos genómicos masivos y desafiantes, incluyendo mamíferos placentarios (240 especies), aves (363 especies), moscas de la fruta (100 especies) y levaduras (332 especies), además de un conjunto más pequeño de bambúes con poliploidía compleja.

Los resultados fueron impresionantes:
- Precisión Comparable al Estado del Arte: Los árboles generados por ROADIES fueron en gran medida consistentes con los publicados en estudios de referencia liderados por expertos, que a menudo requieren meses o años de trabajo manual y análisis detallados. Por ejemplo, en el desafiante conjunto de datos de aves, donde las relaciones evolutivas son notoriamente difíciles de resolver, ROADIES produjo una filogenia altamente congruente con los estudios más recientes.
- Eficiencia Sobresaliente: ROADIES requirió solo una fracción del tiempo y, fundamentalmente, ninguna intervención humana en comparación con los enfoques tradicionales.
- Manejo de Complejidad: Demostró ser robusto incluso en escenarios con alta discordancia entre árboles de genes (como en aves) y poliploidía (como en bambúes).
- Supera a Otros Pipelines Automatizados: Cuando se comparó con otras herramientas más simplificadas o semi-automatizadas (como MashTree, Read2Tree o pipelines basados en BUSCO), ROADIES consistentemente ofreció una mayor precisión, especialmente en filogenias complejas.
¿Para Qué Sirve ROADIES? Democratizando la Filogenómica
La utilidad de ROADIES es vasta y apunta a transformar cómo se realizan los estudios evolutivos:
- Investigadores No Expertos: Permite a biólogos con experiencia limitada en bioinformática compleja generar árboles de especies robustos a partir de sus propios datos genómicos, o de la creciente cantidad de genomas disponibles públicamente.
- Estudios a Gran Escala: Su escalabilidad lo hace ideal para los proyectos genómicos masivos actuales que buscan secuenciar miles de especies (por ejemplo, el Earth BioGenome Project).
- Resolución de Ramas Difíciles del Árbol de la Vida: Al poder muestrear una gran cantidad de regiones genómicas, incluyendo las no codificantes (que pueden tener diferentes señales evolutivas), ROADIES puede ayudar a resolver relaciones evolutivas que han sido contenciosas.
- Generación de Árboles Guía: Puede usarse para generar árboles guía de alta calidad necesarios para herramientas más complejas, como los alineadores de genomas completos.
- Aplicaciones en Biología Comparada y Medicina: Un árbol de especies preciso es fundamental para estudios de genómica comparada, adaptación, evolución de enfermedades, descubrimiento de fármacos y conservación.
Conclusión: Un Salto Adelante para la Filogenómica
ROADIES representa un avance significativo en el campo de la filogenómica. Al automatizar y simplificar drásticamente el proceso de inferencia de árboles de especies a partir de genomas crudos, sin sacrificar la precisión, esta herramienta tiene el potencial de acelerar descubrimientos en una amplia gama de disciplinas biológicas. Al eliminar la necesidad de anotaciones detalladas, manejo de ortología y la dependencia de genomas de referencia, ROADIES no solo ahorra tiempo y recursos, sino que también “democratiza” la capacidad de realizar análisis filogenómicos de vanguardia, poniéndolos al alcance de una comunidad científica mucho más amplia.
Referencia del Artículo:
Gupta, A., Mirarab, S., & Turakhia, Y. (2025). Accurate, scalable, and fully automated inference of species trees from raw genome assemblies using ROADIES. PNAS, 122(19), e2500553122. https://doi.org/10.1073/pnas.2500553122
AI bacterias Biología Sintética CRISPR cáncer envejecimiento Escherichia coli evolución Microbioma microbiota regulación resistencia salud VIH virus
Ultimos Productos
-
Curso de Organismos Modelo en Biología Molecular Febrero 2025
El precio original era: $75.00.$57.00El precio actual es: $57.00. -
Masterclass: Dinámica y Composición de los Condensados Biomoleculares
El precio original era: $80.00.$60.00El precio actual es: $60.00.
Buscar
Últimos Posts
Últimos Comentarios
Categorías
Archivos
- marzo 2026 (5)
- febrero 2026 (15)
- enero 2026 (21)
- diciembre 2025 (20)
- noviembre 2025 (16)
- octubre 2025 (21)
- septiembre 2025 (17)
- agosto 2025 (17)
- julio 2025 (19)
- junio 2025 (24)
- mayo 2025 (26)
- abril 2025 (49)
- marzo 2025 (20)
- febrero 2025 (10)
- enero 2025 (7)
- diciembre 2024 (8)
- noviembre 2024 (23)
- octubre 2024 (7)
Palabras clave
Sígue las noticias
Te invitamos a registrar tus datos como tu correo electrónico para que puedas recibir las últimas noticias y anuncios de Biología Molecular México

By signing up, you agree to the our terms and our Privacy Policy agreement.