¡Tu carrito está actualmente vacío!
Una Nueva Frontera en la Predicción de Interacciones Proteicas
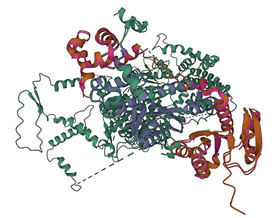
La interacción entre proteínas es fundamental para los procesos biológicos. Desde la formación de estructuras celulares hasta la regulación de señales bioquímicas, las interacciones proteína-proteína (PPIs, por sus siglas en inglés) son esenciales para la vida. Sin embargo, la identificación y caracterización estructural de estas interacciones ha sido un desafío en la biología molecular. El reciente estudio publicado en Molecular Cell [1], presenta Predictomes, una base de datos curada por clasificadores de interacciones proteicas modeladas por AlphaFold, que marca la creación de una nueva forma de usar AlphaFold2 y su función para predecir interacciones entre proteínas, poniendo de manifiesto una nueva forma de evaluar PPIs a gran escala.
Desafíos en el Estudio de las Interacciones Proteicas
El genoma humano codifica alrededor de 20,000 proteínas (que esto lo hemos discutido en este espacio, probablemente son más), lo que genera teóricamente más de 200 millones de combinaciones binarias posibles de interacciones. Sin embargo, solo alrededor de 50,000 interacciones han sido identificadas experimentalmente, y menos de 10,000 han sido resueltas a nivel estructural. Esto evidencia una enorme brecha en nuestro conocimiento sobre el interactoma humano, que es esencial para muchas aplicaciones biomédicas.
Los enfoques experimentales tradicionales, como la co-inmunoprecipitación, la espectrometría de masas (haciendo algunos trucos pequeños para lograr evaluar los pares de interacción) y los ensayos de doble híbrido en bacterias y levaduras, han proporcionado valiosa información sobre las interacciones proteicas. No obstante, estos métodos son laboriosos, costosos y generan tanto falsos positivos como falsos negativos. Con la llegada de AlphaFold2-Multimer (AF-M), un modelo de inteligencia artificial que hemos cubierto bastante en Biología Molecular México, que es capaz de predecir la estructura de complejos proteicos, se ha abierto una nueva vía para la identificación sistemática de PPIs, con el problema de que las métricas de calidad de los modelos no son totalmente fiables para descartar interacciones falsas entre proteínas, de ahí la importancia de este estudio.
Predictomes: Una Solución Basada en Inteligencia Artificial
A pesar del avance que representa AF-M, su aplicación en la predicción de interacciones proteicas enfrenta un problema crítico: la dificultad de distinguir entre interacciones funcionalmente relevantes y predicciones erróneas. Para abordar este desafío, los autores desarrollaron SPOC (Structure Prediction and Omics-informed Classifier), un clasificador basado en aprendizaje automático que mejora la discriminación entre PPIs verdaderas y falsos positivos usando una métrica basada en separar interacciones correctas de no correctas usando la métrica de calidad de los modelos generados y entrenar el modelo con interacciones conocidas contra negativas.

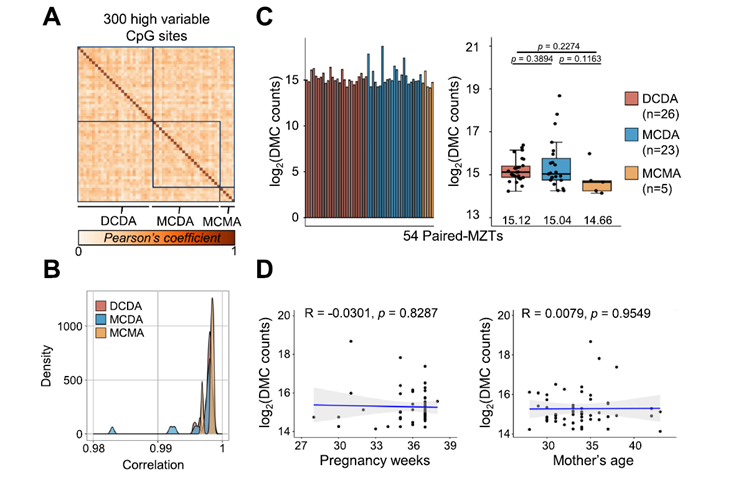
El estudio empleó un análisis de 40,000 predicciones de interacciones en un conjunto de 300 proteínas humanas relacionadas con el mantenimiento del genoma. SPOC utiliza métricas estructurales y datos biológicos de diversos repositorios (como co-expresión, dependencia funcional en células cancerosas y localización subcelular) para asignar una puntuación de confianza a cada interacción.
Impacto y Aplicaciones en la Biología Molecular
La base de datos Predictomes (disponible en predictomes.org) permite a los investigadores explorar interacciones proteicas con alto grado de confianza, formulando nuevas hipótesis para estudios experimentales. Este recurso tiene implicaciones en diversas áreas de la biología molecular, tales como:
– Mantenimiento del Genoma: La identificación de interacciones clave en procesos de replicación y reparación del ADN puede revelar nuevas estrategias para entender y tratar enfermedades relacionadas con inestabilidad genómica.
– Descubrimiento de blancos terapéuticos: La predicción de interacciones entre proteínas patogénicas y humanas puede acelerar el desarrollo de fármacos dirigidos a bloquear interacciones críticas en enfermedades como el cáncer o infecciones virales.
– Biotecnología y Diseño de Proteínas: La posibilidad de predecir interacciones con alta confiabilidad podría optimizar el diseño de complejos proteicos para aplicaciones industriales y terapéuticas.
Conclusión
El desarrollo de Predictomes y el clasificador SPOC representa un avance significativo en la biología estructural y el estudio de interacciones proteicas. Este recurso no solo proporciona una base de datos accesible para la investigación, sino que también establece un marco metodológico para la interpretación de predicciones de AF-M a gran escala. En un futuro cercano, herramientas como SPOC podrían integrarse con métodos experimentales para validar y expandir nuestro conocimiento sobre la organización funcional del proteoma humano.
Referencia [1] Schmid EW, Walter JC. Predictomes, a classifier-curated database of AlphaFold-modeled protein-protein interactions. Mol Cell. 2025 Feb 19:S1097-2765(25)00105-4. doi: 10.1016/j.molcel.2025.01.034.
AI bacterias Biología Sintética CRISPR cáncer envejecimiento Escherichia coli evolución Microbioma microbiota regulación resistencia salud VIH virus
Ultimos Productos
-
Curso de Organismos Modelo en Biología Molecular Febrero 2025
El precio original era: $75.00.$57.00El precio actual es: $57.00. -
Masterclass: Dinámica y Composición de los Condensados Biomoleculares
El precio original era: $80.00.$60.00El precio actual es: $60.00.
Buscar
Últimos Posts
Últimos Comentarios
Categorías
Archivos
- marzo 2026 (5)
- febrero 2026 (15)
- enero 2026 (21)
- diciembre 2025 (20)
- noviembre 2025 (16)
- octubre 2025 (21)
- septiembre 2025 (17)
- agosto 2025 (17)
- julio 2025 (19)
- junio 2025 (24)
- mayo 2025 (26)
- abril 2025 (49)
- marzo 2025 (20)
- febrero 2025 (10)
- enero 2025 (7)
- diciembre 2024 (8)
- noviembre 2024 (23)
- octubre 2024 (7)
Palabras clave
Sígue las noticias
Te invitamos a registrar tus datos como tu correo electrónico para que puedas recibir las últimas noticias y anuncios de Biología Molecular México

By signing up, you agree to the our terms and our Privacy Policy agreement.