¡Tu carrito está actualmente vacío!
El Legado Genético Contra el VIH: Rastrean el Origen Milenario de la Mutación CCR5delta32 en el ADN Antiguo
Una Mutación Providencial en la Lucha Contra el VIH
En la intrincada danza entre nuestros genes y las enfermedades, pocas variantes genéticas han capturado tanto la atención como la deleción CCR5delta32. Esta pequeña alteración de 32 pares de bases en el gen CCR5 confiere una notable resistencia a la infección por el VIH-1, el virus causante del SIDA, especialmente en individuos que heredan dos copias de la mutación. Curiosamente, esta variante se encuentra en frecuencias relativamente altas (10-16%) en poblaciones europeas, un fenómeno que ha desconcertado a los científicos durante décadas. ¿Cuándo surgió? ¿Por qué se volvió tan común? ¿Qué fuerzas evolutivas la impulsaron? Un nuevo estudio publicado en la prestigiosa revista Cell, liderado por investigadores de la Universidad de Copenhague, entre otras instituciones, utiliza la poderosa combinación de genomas antiguos y modernos, junto con herramientas analíticas innovadoras, para trazar la fascinante historia evolutiva de CCR5delta32, revelando orígenes mucho más antiguos y presiones selectivas más tempranas de lo que se pensaba.

Más Allá de la Deleción: Descubriendo los “Barrios” Genéticos (Haplotipos)
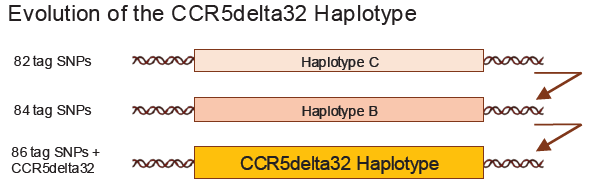
El equipo de investigación no se limitó a buscar la deleción CCR5delta32 de forma aislada. Comprendieron que las variantes genéticas no existen en el vacío, sino que están incrustadas en “barrios” genéticos más grandes llamados haplotipos: secuencias de ADN que tienden a heredarse juntas. Al analizar miles de genomas modernos, identificaron que la deleción CCR5delta32 casi siempre se encuentra en un haplotipo específico, al que llamaron Haplotipo A, caracterizado por 86 variantes genéticas (SNPs) particulares. Descubrieron también otros dos haplotipos relacionados en la misma región del cromosoma 3: el Haplotipo B, que comparte 84 de estas variantes con el A pero no tiene la deleción, y el Haplotipo C, con 82 variantes del B. Este hallazgo fue crucial, sugiriendo que la deleción CCR5delta32 surgió en un Haplotipo B preexistente.

Desenterrando el Pasado: Una Herramienta para el ADN Antiguo (HAPI)
Estudiar deleciones en ADN antiguo es un desafío. El ADN antiguo suele estar muy degradado, fragmentado y presente en bajas cantidades. Para superar esto, los investigadores desarrollaron un ingenioso modelo probabilístico llamado HAPI (Haplotype-Aware Probabilistic model for Indels). HAPI utiliza la información de los SNPs “etiqueta” (tag SNPs) del haplotipo como una “pista” (prior) para ayudar a identificar la presencia de la deleción CCR5delta32 en genomas antiguos de baja cobertura, mejorando significativamente la precisión en comparación con métodos anteriores.

Un Viaje en el Tiempo y el Espacio: Los Verdaderos Orígenes
Armados con HAPI, los científicos analizaron 934 genomas antiguos y más de 2,500 modernos. Sus resultados reescriben la historia de CCR5delta32:
- Origen Antiguo: La deleción CCR5delta32 surgió en el Haplotipo A (derivado del B) hace al menos 6,700 años antes del presente (BP), en la región de la Estepa de Eurasia Occidental. Esto es mucho más antiguo que las estimaciones previas que la situaban entre 700 y 5,000 años atrás, y descarta teorías que atribuían su auge a eventos más recientes como la Peste Negra medieval o las incursiones vikingas.
- Selección Positiva Temprana: El estudio presenta fuerte evidencia de que CCR5delta32 estuvo bajo selección positiva (es decir, fue activamente favorecida por la evolución) entre hace 8,000 y 2,000 años BP, durante el Neolítico Tardío y la Edad del Bronce. Este periodo fue testigo de cambios masivos en la sociedad humana: la transición a la agricultura, el aumento de la densidad poblacional y la domesticación de animales, lo que pudo haber facilitado la aparición y propagación de nuevos patógenos.
- Propagación y Legado: Después de este periodo de selección, la frecuencia del alelo se estabilizó en torno al 10% en Europa. También se detectó la presencia del haplotipo en América Latina, explicable por los intercambios genéticos postcolombinos.

¿Qué Impulsó su Ascenso? Misterios y Pistas
Si bien el estudio no identifica el patógeno exacto que impulsó la selección de CCR5delta32, su antigüedad apunta a presiones selectivas mucho antes de la Peste Negra o la viruela en sus formas más conocidas. Los investigadores sugieren que la ventaja conferida por CCR5delta32, posiblemente relacionada con la modulación de la respuesta inmune a través de los receptores de quimiocinas (CCR2, CCR3, CCRL2, también presentes en el haplotipo), fue crucial durante una época de grandes transformaciones y emergencias de nuevas enfermedades infecciosas. De hecho, el análisis estratificado por ancestralidad mostró que la selección fue particularmente fuerte en poblaciones con ascendencia de Cazadores-Recolectores del Este (EHG) y del Cáucaso (CHG).
Implicaciones y Relevancia Actual
Los hallazgos de este estudio tienen profundas implicaciones:
- Contexto Médico Ampliado: Entender que CCR5delta32 es parte de un haplotipo más grande es vital. Las otras variantes genéticas en el Haplotipo A podrían tener sus propios efectos sobre la salud o modular el efecto de la deleción. Esto debe considerarse al desarrollar terapias que apunten al receptor CCR5, no solo para el VIH sino para otras condiciones donde está implicado (enfermedades autoinmunes, cáncer).
- Precaución con los Marcadores: El estudio advierte que usar un único SNP “etiqueta” para inferir la presencia de CCR5delta32 puede ser erróneo, dada la compleja estructura de haplotipos y la presencia de variantes similares en haplotipos sin la deleción, especialmente en poblaciones no europeas.
- Historia de Adaptación Humana: Proporciona una ventana detallada a cómo las poblaciones humanas se adaptaron a las presiones patogénicas en el pasado profundo, mucho antes de las epidemias históricamente documentadas.
Conclusión: Un Retrato Detallado de Nuestra Evolución Inmunitaria
El trabajo de Ravn y colaboradores es un tour de force que combina la paleogenómica, la genética de poblaciones y la bioinformática avanzada para pintar el retrato más completo hasta la fecha de la historia evolutiva de la variante CCR5delta32. Al revelar su origen antiguo en la Estepa Euroasiática y el intenso periodo de selección positiva durante el Neolítico y la Edad del Bronce, este estudio no solo resuelve un debate de larga data, sino que también subraya la importancia de considerar el contexto haplotípico completo de las variantes genéticas con relevancia médica. Es un recordatorio de que nuestro genoma es un archivo histórico viviente, con historias de supervivencia y adaptación grabadas en nuestro ADN a lo largo de milenios.
Referencia: Ravn, K., Cobuccio, L., Muktupavela, R. A., Meisner, J., Danielsen, L. S., Benros, M. E., Korneliussen, T. S., Sikora, M., Willerslev, E., Allentoft, M. E., Irving-Pease, E. K., & Rasmussen, S. (2025). Tracing the evolutionary history of the CCR5delta32 deletion via ancient and modern genomes. Cell, 188, 1–17. https://doi.org/10.1016/j.cell.2025.04.015 (Nota: La fecha de publicación indicada es “July 10, 2025”, lo que sugiere que es un artículo aceptado para publicación futura o una “preview”).
AI bacterias Biología Sintética CRISPR cáncer envejecimiento Escherichia coli evolución Microbioma microbiota regulación resistencia salud VIH virus
Ultimos Productos
-
Curso de Organismos Modelo en Biología Molecular Febrero 2025
El precio original era: $75.00.$57.00El precio actual es: $57.00. -
Masterclass: Dinámica y Composición de los Condensados Biomoleculares
El precio original era: $80.00.$60.00El precio actual es: $60.00.
Buscar
Últimos Posts
Últimos Comentarios
Categorías
Archivos
- marzo 2026 (5)
- febrero 2026 (15)
- enero 2026 (21)
- diciembre 2025 (20)
- noviembre 2025 (16)
- octubre 2025 (21)
- septiembre 2025 (17)
- agosto 2025 (17)
- julio 2025 (19)
- junio 2025 (24)
- mayo 2025 (26)
- abril 2025 (49)
- marzo 2025 (20)
- febrero 2025 (10)
- enero 2025 (7)
- diciembre 2024 (8)
- noviembre 2024 (23)
- octubre 2024 (7)
Palabras clave
Sígue las noticias
Te invitamos a registrar tus datos como tu correo electrónico para que puedas recibir las últimas noticias y anuncios de Biología Molecular México

By signing up, you agree to the our terms and our Privacy Policy agreement.